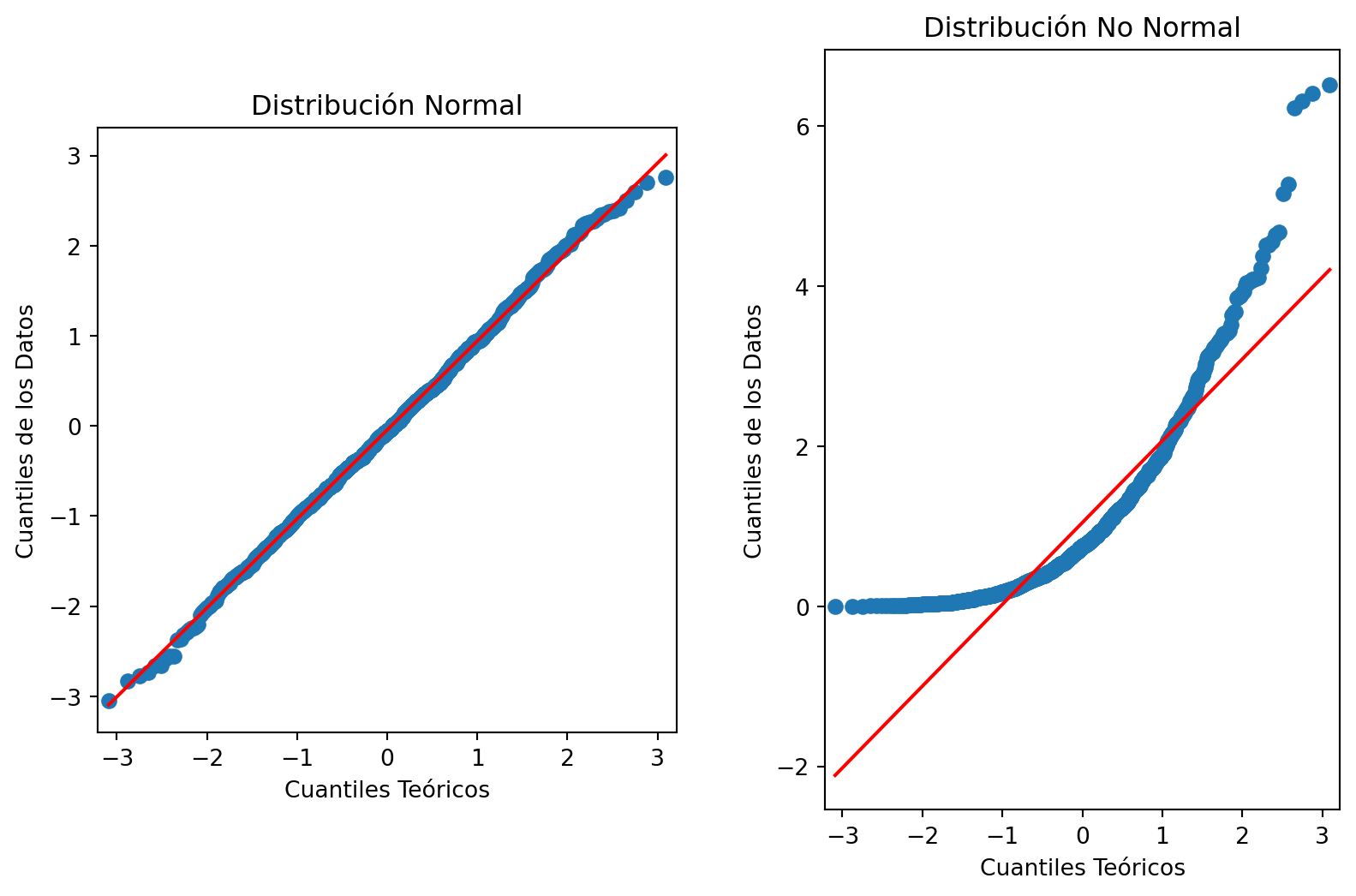
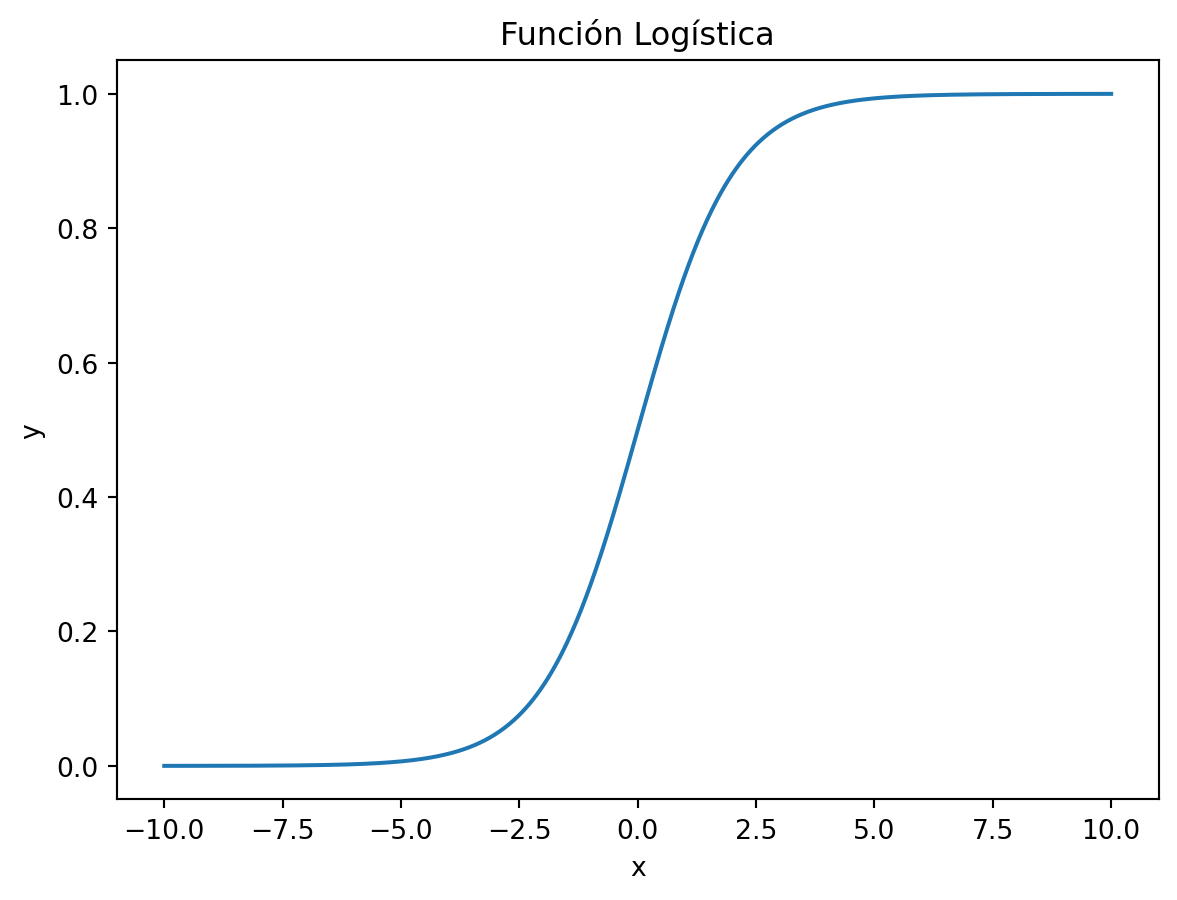
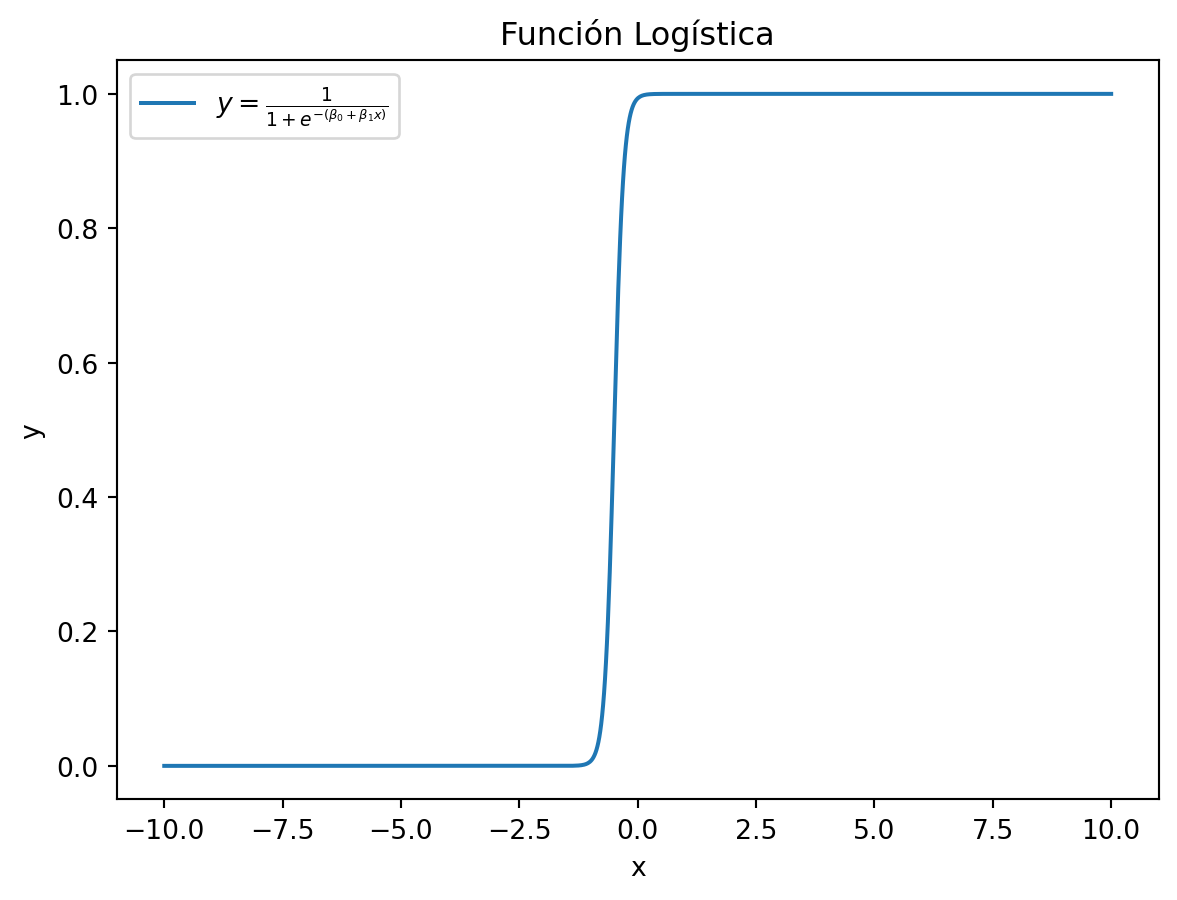
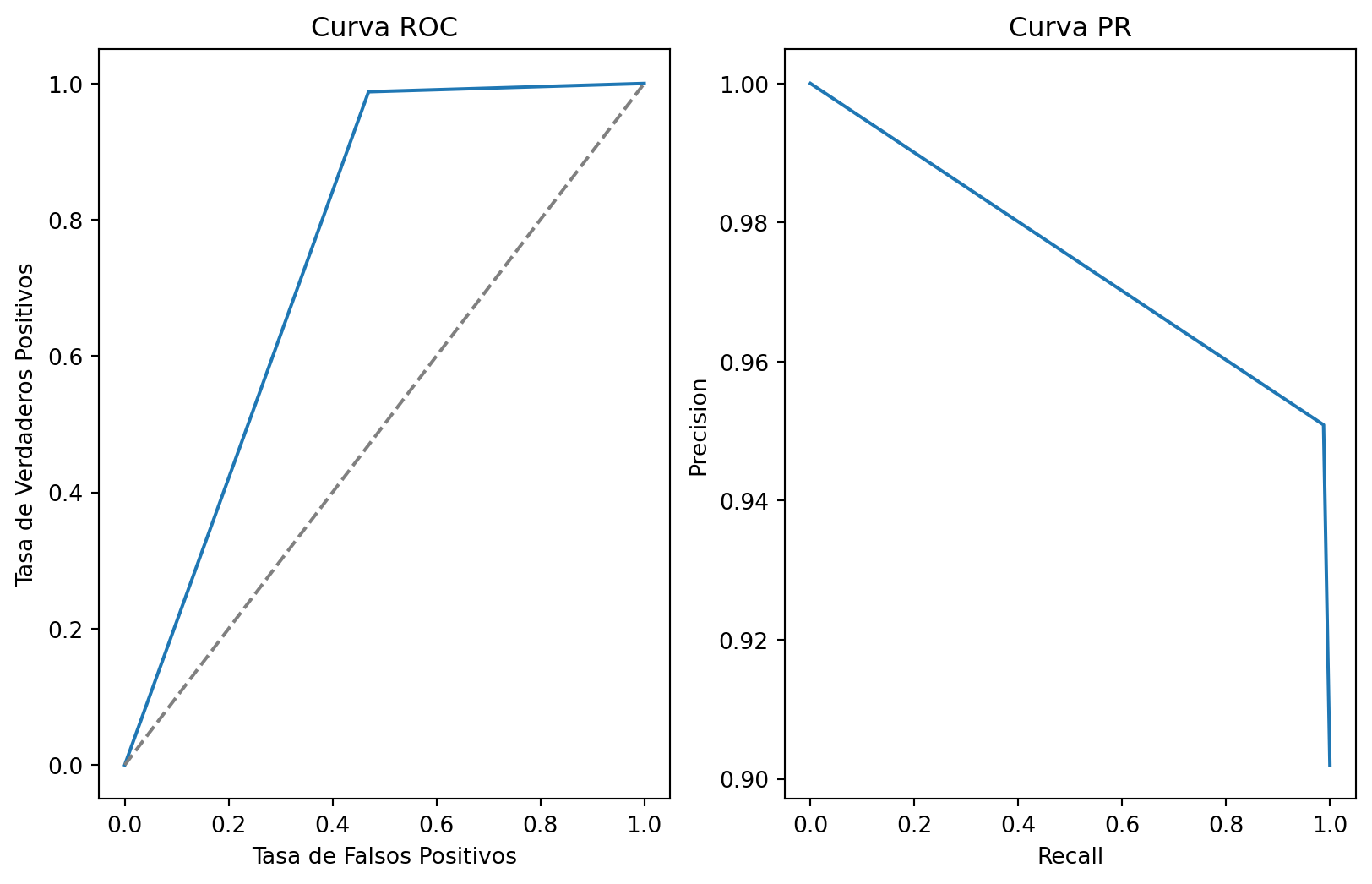
array([[ 1. , 187.64052346],
[ 1. , 174.00157208],
[ 1. , 179.78737984],
[ 1. , 192.40893199],
[ 1. , 188.6755799 ],
[ 1. , 160.2272212 ],
[ 1. , 179.50088418],
[ 1. , 168.48642792],
[ 1. , 168.96781148],
[ 1. , 174.10598502],
[ 1. , 171.44043571],
[ 1. , 184.54273507],
[ 1. , 177.61037725],
[ 1. , 171.21675016],
[ 1. , 174.43863233],
[ 1. , 173.33674327],
[ 1. , 184.94079073],
[ 1. , 167.94841736],
[ 1. , 173.13067702],
[ 1. , 161.45904261],
[ 1. , 144.47010184],
[ 1. , 176.53618595],
[ 1. , 178.64436199],
[ 1. , 162.5783498 ],
[ 1. , 192.69754624],
[ 1. , 155.45634325],
[ 1. , 170.45758517],
[ 1. , 168.1281615 ],
[ 1. , 185.32779214],
[ 1. , 184.6935877 ],
[ 1. , 171.54947426],
[ 1. , 173.7816252 ],
[ 1. , 161.12214252],
[ 1. , 150.19203532],
[ 1. , 166.52087851],
[ 1. , 171.56348969],
[ 1. , 182.30290681],
[ 1. , 182.02379849],
[ 1. , 166.12673183],
[ 1. , 166.97697249],
[ 1. , 159.51447035],
[ 1. , 155.79982063],
[ 1. , 152.93729809],
[ 1. , 189.50775395],
[ 1. , 164.90347818],
[ 1. , 165.61925698],
[ 1. , 157.4720464 ],
[ 1. , 177.77490356],
[ 1. , 153.86102152],
[ 1. , 167.8725972 ],
[ 1. , 161.04533439],
[ 1. , 173.86902498],
[ 1. , 164.89194862],
[ 1. , 158.19367816],
[ 1. , 169.71817772],
[ 1. , 174.28331871],
[ 1. , 170.66517222],
[ 1. , 173.02471898],
[ 1. , 163.65677906],
[ 1. , 166.37258834],
[ 1. , 163.27539552],
[ 1. , 166.40446838],
[ 1. , 161.86853718],
[ 1. , 152.73717398],
[ 1. , 171.77426142],
[ 1. , 165.98219064],
[ 1. , 153.69801653],
[ 1. , 174.62782256],
[ 1. , 160.92701636],
[ 1. , 170.51945396],
[ 1. , 177.29090562],
[ 1. , 171.28982911],
[ 1. , 181.39400685],
[ 1. , 157.6517418 ],
[ 1. , 174.02341641],
[ 1. , 163.15189909],
[ 1. , 161.29202851],
[ 1. , 164.21150335],
[ 1. , 166.88447468],
[ 1. , 170.56165342],
[ 1. , 158.34850159],
[ 1. , 179.00826487],
[ 1. , 174.6566244 ],
[ 1. , 154.63756314],
[ 1. , 184.88252194],
[ 1. , 188.95889176],
[ 1. , 181.78779571],
[ 1. , 168.20075164],
[ 1. , 159.29247378],
[ 1. , 180.54451727],
[ 1. , 165.96823053],
[ 1. , 182.2244507 ],
[ 1. , 172.08274978],
[ 1. , 179.76639036],
[ 1. , 173.56366397],
[ 1. , 177.06573168],
[ 1. , 170.10500021],
[ 1. , 187.85870494],
[ 1. , 171.26912093],
[ 1. , 174.01989363]])